3.5 Funciones
El tema de las funciones es algo extenso, por lo que se dará de manera resumida el contenido necesario para entender gran parte del funcionamiento de estas en R. Una de las mejores fuentes de información la pueden encontrar en el libro Advance R, específicamente en los capítulos 6 y 7; para explorar más sobre este tema se puede consultar la segunda parte de la misma referencia. Finalmente, se verá como usar vectorización, una de las fuertes habilidades de R, y por último una serie de funciones que podrían ser de gran utilidad en ciertos temas.
Particularmente en la programación con R, será de mucha utilidad aprender a crear funciones:
funcionfactorial<-function(a){
res <- 1
if(a<0){
return("No existen factoriales de números negativos.")
} else if(a==0){
return(res)
} else{
for(i in 1:a){
res <- res*i
}
return(res)
}
}3.5.1 Funciones
Una función que crea el usuario se compone de tres partes: los formales, los cuales son los argumentos o los parámetros de una función y que pueden obtenerse con la función formals(), el cuerpo, es decir el código o las rutinas internas de las funciones que se pueden obtener con la función body(), y un ambiente, el cual se puede obtener con la función environment() y que básicamente es un lugar donde esta almacenada la información necesaria para el correcto uso de las funciones; más adelante se hablará un poco más de este importante concepto.
- ¿Qué tipo de estructuras o qué tipo de objetos regresan las anteriores funciones?
- ¿Cuáles son los tres componentes en la siguiente función?
myStrangeFunction <- function(a = 1,b = 4,c,...) sum(prod(a,b), c, ...)Como ya se mencionó, este lenguaje toma ciertas características de un lenguaje funcional, por lo que al tratar a las funciones como objetos es de esperarse la incorporación de atributos a estas. Uno de estos atributos es el llamado srcref (source reference), la cual imprime mejor el cuerpo de alguna función.
- ¿Con qué función se agregaría un atributo a una función?
- ¿Cómo se puede obtener el atributo srcref de
myStrangeFunction? El output es el siguiente
function(a = 1,b = 4,c,...) sum(prod(a,b), c, ...)R contiene ciertas funciones que son añadidas al momento de iniciar su interfaz, las cuales se han agregado a las distintas versiones del lenguaje y a medida que R ha evolucionado se han optimizado estas funciones internas dando un buen rendimiento para su uso cotidiano. Estas funciones son llamadas primitivas y ya se han visto algunas de estas: sum(), prod(), [(), sqrt(), etc. Algunas de las funciones primitivas están implementadas y existen en el lenguaje C, lo cual da garantía de un buen rendimiento. Los tres componentes que se mencionaron antes no los tienen este tipo de funciones y así es posible verificar si una función primitiva existe principalmente en C.
- ¿Cómo se puede ver si una función es primitiva o no?
- Si las funciones primitivas otorgan un buen rendimiento ¿Porqué no todas las funciones son primitivas?
function (..., na.rm = FALSE) .Primitive("sum")Para utilizar, o mejor dicho llamar, a una función, basta con escribir su nombre, colocar los argumentos separados por coma dentro de unos paréntesis y ¡listo! Aunque se ha mencionado que + también es una función: '+'(), por lo que hay que discernir las diferentes formas de llamar a una función.
- prefix:
myFunction(arguments). - infix:
a+b. - replacement:
colnames(df) <- names. - special:
[[,if,for, etc.
Véase que las propias estructuras de control y los bucles también son funciones y R permite modificar todo aquello que sea función, aunque todas las formas especiales son funciones primitivas. No es recomendable sobre escribir alguna función que ya se tenga preestablecida en R o en alguna librería, pero siempre se tiene la libertad de hacerlo en caso de que se tenga el control y la consciencia suficiente de las consecuencias.
Solo para aclarar, véase los siguientes puntos.
- Todas las funciones se pueden llamar a manera de prefijo.
myFunction(a,b,c)
`+`(a,b)
`colnames<-`(df, names)
for(name in names) print(name)
`for`(name, names, print(name))- Como
+es una función, es posible crear operadores usando%al inicio y al final del operador. Todos los operadores de R se pueden encontrar ejecutando?Syntax.
`%mixOperations%` <- function(a,b) a+b*a/b
4 %mixOperations% 5[1] 8Algunas funciones útiles de este estilo son
%%,%*%,%/%,%in%,%o%y%x%. ¿Qué hacen cada una de ellas?Es análogo la sintaxis para crear funciones de asignación aunque es necesario tener el formal
value.
`indexNames<-` <- function(a, value) {
names(a) <- as.character(seq_along(a)+value)
a
}
x <- 1:20
indexNames(x) <- 3
x 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 Ahora, véase la siguiente función
ignoringParameters <- function(x) "Hello!"
ignoringParameters(1:30)[1] "Hello!"Sea cual sea el objeto que se de como parámetro al llamar la función, se devolverá el mismo resultado. Esto es posible porque las funciones en R trabajan de manera “perezosa” con sus argumentos, a esto se le llama lazy evaluation. Básicamente, si no es llamado en algún momento un argumento de la función, este no es utilizado y solo cuando se llame al argumento, hasta ese momento se hará el acceso a él.
Esto es gracias a la técnica de evaluación lazy evaluation que utiliza R al usar una estructura de datos llamada promise donde, básicamente las variables son evaluadas mediante un ambiente hasta que una expresión las utilice y este valor es guardado (cached), por ejemplo
y <- 7
lazilyEvaluated <- function(x, lazy_var = y*a){
print("Hi!")
y <- 50
a <- 3
print(lazy_var)
x*2
}
doubleVector <- function(x) c(x,x)
doubleVector(lazilyEvaluated(y))[1] "Hi!"
[1] 150[1] 14 14- Dentro del ambiente de la función, el nombre de la variable
ytiene asociado un valor distinto y no tiene consciencia de la variable con el nombreyfuera de él. - Al utilizar la variable
ycomo argumento de la función, a esta se le da el nombre dexpero se le asocia el mismo valor dey. - El parámetro
lazy_vares llamado hastaprint(lazy_var), donde antes se asignaron valores a las variablesyya, dando así una correcta evaluación alazy_var; es decir que hasta ese momento fue necesario evaluarlazy_varpor lo cual no se genera algún problema. - ¿Por qué sólo se imprimió una vez el
"Hi!"y el valor delazy_var? Por el valor calculado y guardado en la promise.
Otra forma de llamar a una función es usando la función do.call(), la cual permite agregar una lista de parámetros.
do.call(myStrangeFunction, args = list(a = 2, b = 3, c = 4, 6,7,8,9))[1] 40Los objetos que guardemos o asignemos en nuestros flujos de trabajo serán almacenados en un espacio de memoria que se llama ambiente o en inglés environment. Desde RStudio podremos ver lo que se guardado en este espacio de memoria en el panel Environment. Este tema es un poco extenso por lo que hay que entender sólo algunas cosas.
- Un ambiente en R es lo que se conoce como una cerradura (closure) en programación.
- Una cerradura es una técnica para almacenar la información necesaria y permitir la integridad de esta en una función.
- Un ambiente se encarga de asociar un conjunto de nombres a un conjunto de valores, por lo que los valores no estan directamente almacenados en el ambiente, si no sólo los enlaces identificados por los correspondientes nombres.
- En un ambiente todos los nombres son únicos y no están ordenados (recordar que los nombres están asociados a las variables o valores para un uso posterior, por ejemplo:
var <- c(1,2)). Para crear un ambiente se utiliza la funciónnew.env(). - La función
ls()permite ver todos los elementos de un ambiente. Se utiliza la funciónassign()para agregar un nuevo elemento a un ambiente dado y para obtener alguno de los elementos se puede acceder con el operando$, como en una lista o con la funciónget()(si no se encuentra la variable con el nombre dado, get buscará en los ambientes padres). Para preguntar si existe alguna variale con un nombre dado en un ambiente se utiliza la funciónexists()y para eliminar una o varias referencias de variables en un ambiente se usa la funciónrm().
env_test <- new.env()
assign("new_variable", 1:5, envir = env_test)
ls(env_test)[1] "new_variable"env_test$new_variable[1] 1 2 3 4 5# get("new_variable", envir = env_test)
exists("new_variable", envir = env_test)[1] TRUErm("new_variable", envir = env_test)
ls(env_test)character(0)Más ejemplos
## Muestra los nombres de los objetos en memoria
ls()
## Muestra las variables con cierta serie de caracteres en su nombre
ls(pat="m")
## Muestra las variables las cuales su nombre empieza con el caracter dado
ls(pat="^m")
## Muestra detalles de los objetos en memoria
ls.str()
## Eliminar todas las variables de 'Global Environment'
rm(list=ls())
## Eliminar únicamente variables que empiezan con la letra m
rm(list=ls(pat="^m"))
## Tipo de elementos del objeto v
mode(v)
## Longitud del objeto v
length(v)- En un ambiente se pueden acceder a las variables que están enlazas en el mismo mediante su nombre, lo cual garantiza que, si dado una variable en otro ambiente con un mismo nombre a alguna variable en el ambiente actual, estas no sean confundidas y se obtengan resultados inesperados.
- Cada ambiente tiene un padre, lo cual permite que existan enlaces entre las librerías, permitiendo la inclusión de funciones en distintos paquetes. Para ver el ambiente padre se utiliza la función
parent.env()dando como parámetro un ambiente. Véase la siguiente imagen
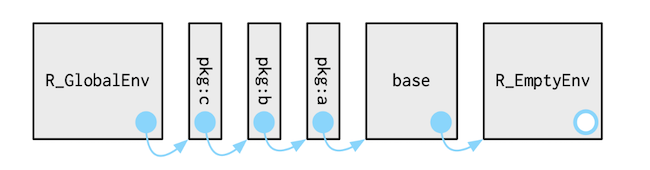
Fuente: Advance R.
El ambiente donde regularmente se trabaja es el R_Global_Environment también llamado workspace. Cada vez que se agrega una nueva librería, el environment para esta (package environment) es colocado entre el ambiente de la última librería cargada y el ambiente global. Para ver todos los ambientes activos de este tipo se puede usar la función search().
- Cada vez que se crea una función, esta es enlazada con el ambiente global mediante un function environment.
- Cuando los ambientes son modificados, estos no son copiados; copiados en el sentido que se verá más adelante en la sección Iteración y Recursión.
- Cada función en un paquete esta asociado con un ambiente del paquete y un ambiente especial llamado namespace environment, el cual contiene todos los enlaces a las variables de la función, y un import environment que contiene todos los enlaces a las funciones que requiera la función del paquete actual.
- Cada vez que se ejecuta una función, se crea un execution environment donde su padre es el ambiente de la función.
Recordando un poco de lo aprendido en programación orientada a objetos, existe la analogía de que las cerraduras son una manera de simular el encapsulamiento en las funciones, y ya que las funciones son de primera clase en R, son tratadas también como objetos.
Para más información acerca de los temas anteriores consúltese Advance R. Second Edition y Advance R. First Edition.
3.5.2 Vectorización
Desde las estructuras de datos es fácil ver la importancia que tienen los vectores en R y la facilidad de usar funciones y operaciones en ellos, logrando tener un buen rendimiento, evitando ciclos y ganando comprensión en los resultados esperados.
La Vectorización significa aplicar funciones optimizadas, en este caso escritas en C, para llegar a una solución del problema a tratar. Esto da soluciones más simples tratando a los vectores como unidad en lugar de pensar en una solución donde se tiene que tratar cada entrada en el vector. En este último caso se tendría que hacer algún tipo de ciclo, en el caso de la vectorización los ciclos son hechos directamente en C, los cuales son más rápidos y con menor sobre carga.
Ya se han tratado algunas funciones vectorizadas, como lo son sum(), prod() y sqrt(), las cuales todas sirven con un vector.
- ¿Qué sucede con
sum(list(1,2,3,4,4,5)),sum(matrix(1,ncol = 3,nrow = 6))ysum(data.frame(x = 1:20, y = 2:21))?
Dado lo anterior, ¿Se puede obtener la suma por renglones o columnas en una matriz? ¿Se puede hacer lo mismo en un data frame o una lista? ¿Se puede aplicar una función particular que trabaje de manera vectorial por renglones o columnas en un data frame? La respuesta a todo esto es ¡Sí! y se puede realizar todo con la familia de funciones apply.
La familia apply es un conjunto de funciones que ayudan en la aplicación vectorial de funciones sobre ciertas estructuras de datos. Para ver a detalle el funcionamiento de cada una de estas y otras funciones, puede consultarse el tutorial de DataCamp Tutorial on the R Apply Family y los enlaces que se dejan al final de esta sub-sección. Mientras tanto, véase los siguientes ejemplos y funciones.
La función apply(X, MARGIN, FUN, ...) ejecuta una función en un arreglo o una matriz X. El atributo MARGIN es utilizado para determinar si la aplicación vectorial será por renglones o columnas (MARGIN = 1 aplicará FUN sobre renglones, MARGIN = 2 sobre columnas y MARGIN=c(1,2) para ambos)
seq_matrix <- matrix(1:12, nrow = 4, ncol = 3)
sum_n_times <- function(x, n) sum(x*n)
apply(seq_matrix, MARGIN = 1, sum)[1] 15 18 21 24apply(seq_matrix, MARGIN = 2, FUN = sum_n_times, 5)[1] 50 130 210En el código anterior se utilizó la función sum_n_times, la cual puede solo usarse para este caso y en ninguna otra ocasión. Esto puede suceder cuando se requiera un resultado especial y único, por lo que declarar y reservar un espacio en memoria para una función y todo lo que implica podría ser un uso inadecuado del espacio disponible. Para estos casos se puede utilizar una función anónima, las cuales son conocidas como funciones lambda.
seq_data_frame <- data.frame(x = 1:12, y = 2:13)
apply(seq_data_frame, MARGIN = 2, FUN = function(x) sum(x*5)) x y
390 450 Véase que se utilizo un data frame en la función apply anterior. Esto funciona pero no es la manera correcta de hacer vectorización con data frames. En otro momento se verá el porqué de esto.
La función lapply acepta arreglos, data frames, vectores y listas y tiene la peculiaridad de regresar una lista.
lapply(list(a = 1:20, b = 2:30), sum)$a
[1] 210
$b
[1] 464lapply(list(a = list(1,2,3), b = list(4,5,6), c = list(7,8,9)), FUN = "[", 2)$a
$a[[1]]
[1] 2
$b
$b[[1]]
[1] 5
$c
$c[[1]]
[1] 8lapply(seq_matrix[c(1,2),c(1,2)], "[", 1)[[1]]
[1] 1
[[2]]
[1] 2
[[3]]
[1] 5
[[4]]
[1] 6lapply(list(a = seq_matrix, b = seq_matrix+1, c = seq_matrix+3), FUN = "[", 1,2)$a
[1] 5
$b
[1] 6
$c
[1] 8- Véase que se utilizo el operador
'['para extraer elementos de las listas y elementos de las matrices. - ¿Qué significa el parámetro 1 en
'[cuando se utilizó paraseq_matrix[c(1,2),c(1,2)]? Hint: Véase la descripción del argumentoXen la documentación delapply(). - ¿Qué significa el parámetro 2 en
'[cuando se utilizó para el segundo ejemplo? - ¿Qué se obtiene en el último ejemplo?
Otra de las funciones más utilizadas es sapply(). Esta función devuelve una salida con la estructura más básica posible, por lo que es común obtener vectores con esta función. sapply() es una versión amigable de lapply() por el tipo de salida, aunque modificando el parámetro simplify a FALSE, sapply tiene el mismo comportamiento que lapply().
sapply(list(a = 1:20, b = 2:30), function(x) sqrt(sum(x))) a b
14.49138 21.54066 sapply(list(a = 1:20, b = 2:30), function(x) sqrt(sum(x)) , simplify = FALSE)$a
[1] 14.49138
$b
[1] 21.54066- Aún falta ver las funciones
tapply(),mapply()yvapply(). - ¿Qué hace
replicate()? - ¿Qué hace
which()?
Aquí se enlistan algunos enlaces útiles sobre este tema:
3.5.3 Paquetes
Antes de ver un compendio de funciones útiles, hay que tener en cuenta que reutilizar código es muy importante para no perder tiempo en la resolución de problemas; y antes de instalar librerías o usar otras funciones base de R, hay que hablar sobre el CRAN.
El CRAN (Comprehensive R Archive Network) es una red de servidores web y FTP (File Trasnfer Protocol) donde se almacenan versiones de código y documentación de R. Además proporciona los hipervínculos para muchos paquetes con diversos temas (se puede consultar todo esto en el CRAN Task Views). Como es difícil e ineficiente tener todo en un mismo lugar, se crearon copias (mirrors) al rededor del mundo donde se mantiene y da soporte a toda esta información. Se puede consultar en la siguiente enlace la lista de todos estos lugares. Para el caso de México, se tienen dos ubicaciones:
Para instalar paquetes se puede elegir alguna de las siguientes opciones:
- Instalación directa:
install.packages("tidyverse")
install.packages("file:///source", repos=NULL)- Mediante RStudio: Tools > Install.Packages …
Regularmente, los paquetes usarán en su codificación funciones de otros paquetes, por lo que será necesario instalar todas los paquetes de los que depende para su correcto uso. Para evitar instalar uno por uno los paquetes de los que dependa alguno, se agrega el argumento dependences=TRUE a la función install.packages(): install.packages(...,dependeces = TRUE).
Lo anterior sirve para aquellos paquetes que están en el CRAN, pero si no lo están o se desea instalar otra versión de los paquetes, se puede usar devtools, el cual permite instalar un paquete directamente desde su fuente, como es el caso de algunos localizados en github.
install.packages("devtools")
library("devtools")
install_github("hadley/emo")Al instalar un paquete, este es guardado en el ordenador; ya que los paquetes no son más que archivos y carpetas que pueden contener funciones en Scripts, archivos .Rsd (Bases de datos de R) entre otros.
Ejemplo para el paquete actuar
La ubicación donde el paquete es guardado se le llama library y para identificar donde empieza la busqueda R al cargar un paquete se utiliza el comando .libPaths(). ¿Qué sucede al ejecutar lapply(.libPaths(), dir)?
Para cargar un paquete se puede utilizar las funciones library() y require. Ambas funciones cargan lo contenido en los paquetes aunque existen pequeñas diferencias entre ambas. Al querer cargar un paquete que no esta instalado, library devolverá un error y require una advertencia, además require regresa un valor booleano dependiendo si se encuentra o no la librería para cargar dicho paquete; esto puede ser útil dentro de funciones para preparar el workspace y sus enlaces. Por ejemplo, en la siguiente función se instala el paquete dplyr en caso de no se tenga la librería o se carga el paquete en caso contrario.
preparation <- function(){
if(!require("dplyr")){
install.packages("dplyr")
}
}Al momento de querer usar una función de algún paquete se puede llamar como cualquier función, aunque puede darse el caso de que existan los mismos nombres entre dos o más librerías para una función o hasta el mismo nombre para una función que creó el usuario. En tal caso se puede especificar el paquete con el operador ::; por ejemplo stats::chisq.test().
- ¿Para qué sirve la función
attach()ydetach?
3.5.4 Miscellaneous
A continuación se dará una serie de funciones y paquetes con los que se puede trabajar en ciertas ocasiones. Aquí no se mencionarán funciones que “vivan” en el “tidyverse” o funciones dedicadas a elementos gráficos, así como funciones relacionadas a temas avanzados de estadística y probabilidad.
3.5.4.1 Vectores
base::cumsum(): Dado un vector, la funcióncumsumdevuelve la suma acumulada de dicho vector. También se tienebase::cummin(),base::cummax()ybase::cumprod().base::unique(): Esta función elimina los elementos duplicados en un vector, data frame o un arreglo.base::sort(): La funciónsortregresa de manera ordenada su input, el cual debe ser un vector o factor.base::gl(): Genera series regulares de factores dados. Ejemplos:gl(3, 5, length = 30),gl(2, 6, label = c("Hombre","Mujer")).base::union(),base::intersect(),base::setdiff()ybase::setequal(): Funciones para realizar operaciones de conjuntos en vectores.package:vctrs: Este paquete ofrece varias funciones útiles en el manejo de vectores, además de que algunas de sus funciones se pueden usar en otro tipo de estructuras de datos.
3.5.4.2 Matrices y Data frames
base::rowSums()ybase::rowMeans(): Con estas funciones se obtienen la suma o el promedio por renglón en una matriz. Existe el equivalente para las columnas y son más rápidas estas funciones que al aplicarapply.- ¿Qué hace
base::rowsum()?
- ¿Qué hace
utils::head()yutils::tail(): Funciones para obtener las primeras o últimas observaciones de un data frame; también funciona con matrices.base::split(): Dicha función separa en grupos de acuerdo a una variable.package:data.table: El paquete data.table contiene varias funciones para manipular data.tables, los cuales pueden obtenerse fácilmente con un data frame. Las funciones en este paquete son eficientes en memoria y muy rápidas.- La función
base::expand.grid()genera un data frame con todas las posible combinaciones de vectores o factores dados como argumentos. Aquí un ejemplo:expand.grid(h = c(60, 80), w = c(100, 300), sex = c("Hombre", "Mujer")).
3.5.4.3 Lectura y escritura
En cuanto a archivos externos, en general se utilizan archivos con extensión .csv y .txt; sin embargo R no está limitado a este tipo de archivos. Existen una amplia variedad de formatos que se pueden leer a través de paquetes diseñados específicamente para ese fin.
El siguiente ejemplo muestra cómo se haría la lectura de un archivo con extensión .txt:
datos<-read.table("data.txt", # nombre del archivo (con extensión) entre comillas
header = TRUE, # TRUE o FALSE, indicando si el archivo tiene como primer renglón el nombre de las columnas
sep="\t" # separador de los campos
)Una vez que leemos el archivo externo en R, el objeto donde se almacenará la información será de tipo data.frame.
Muchas veces será de nuestro interés exportar objetos data.frame a archivos externos, tal objetivo lo podemos lograr con alguna de las siguientes dos opciones:
write.table(datos, "toma.txt", append=F, sep="\t")
write.csv(datos, "toma2.csv")N:B. Como en la lectura de datos, R no está limitado a exportar archivos .csv o .txt también existe una amplia variedad de formatos que podemos generar. En la siguiente lista se aclaran algunas funciones utilizadas y se añaden otras que pueden ser de interés.
base::read.csv(),base::read.delim(),base::read.table()ybase::readLines(): Diferentes funciones para leer archivos de texto en distintos formatos.base::write(),base::csv()ybase::table(): Funciones para exportar datos, como un data frame o una matriz, en un formato especifico.base::saveRDS(): Función para exportar o guardar algún objeto de R. Se puede leer este objeto con la funciónbase::readRDS(). Estas funciones no están optimizadas y existen algunas variantes para estas funciones comoreadr::save_rdsyqs::qsave(). Aún así, este tipo de almacenamiento de datos (en forma de bytes) es de lo más confiable ya que es serializable. Se recomienda leer el post A better way of saving and loading objects in R.
3.5.4.4 Funciones estadísticas y matemáticas
base::sample(): Función para obtener una muestra aleatoria de \(n\) números y permutaciones de un conjunto dado.set.seed(): Función para asignar la semilla para los métodos pseudo aleatorios posteriores.base::sd(): desviación estándar.base::var(): varianza.base::min(): mínimo.base::max(): máximo.base::median(): mediana.base::range(): rango.base::quantile(): cuantiles.
set.seed(20)
data <- data.frame(v = sample(c(1:25, NA), size = 50, replace = T),
w = sample(c(1:25, NA), size = 50, replace = T),
x = sample(c(1:25, NA), size = 50, replace = T),
y = sample(c(1:25, NA), size = 50, replace = T),
z = sample(c(1:25, NA), size = 50, replace = T))
head(data) v w x y z
1 6 1 17 NA 13
2 11 15 8 11 22
3 24 6 3 15 21
4 2 2 11 8 20
5 25 6 7 3 19
6 2 10 18 25 24data.frame(Sd = sapply(data, sd, na.rm = T),
Varianza = sapply(data, var, na.rm=TRUE),
Min = sapply(data, min, na.rm=TRUE),
Max = sapply(data, max, na.rm=TRUE),
Mediana = sapply(data, median, na.rm=TRUE),
MissingValues = sapply(data, function(x) sum(is.na(x)))) Sd Varianza Min Max Mediana MissingValues
v 7.359459 54.16163 2 25 14.0 0
w 7.845289 61.54857 1 25 8.0 3
x 7.646861 58.47449 1 25 10.0 1
y 7.126090 50.78116 1 25 11.0 4
z 7.176597 51.50355 1 25 14.5 2Lo anterior es un pequeño resumen cuantitativo de los datos generados, pero se pueden obtener con diferentes funciones como base::sumary(), base::fivenum() y Hmisc::describe().
Con los paquetes
naniaryvisdatase puede dar tratamiento a los valores perdidos, así como elementos gráficos de resumen sobre estos.base::abs(): Valor absoluto.base::sqrt(): Raíz cuadrada.base::exp(): Función exponencial.base::log(): Logaritmo Natural.base::log2(),base::log10(),base::log(base): Logaritmo en diferente base.base::sin(),base::cos(),base::tan(),base::asin(),..: Funciones trigonométricas.base::integrate(): Función para encontrar el área debajo de una curvabase::uniroot(): Función para encontrar las raíces de una función.base::optimse(): Función para encontrar el mínimo o máximo de una función.base::lfactorial(): Esta función calcular el logaritmo natural del factorial de un número.base::cut(): Dicha función establece intervalos en todo el rango de valores dados por un vector. ¿El resultado es un factor?base::findInterval(): Misma finalidad de la funcióncutpero es más rápido que dicha función.
3.5.4.5 Others
base::object.size(): Esta función regresa una aproximación de la memoria utilizada por un objeto.base::capture.output(): Dicha función captura la salida de una función en un caracter.base::readlines(): Lectura de una línea que el usuario ingresa desde la terminal.base::invisible()ybase::withVisible(): En ciertas ocasiones se desea que el resultado de las funciones no sea impreso, en tal case se utiliza la funcióninvisibley para poder visualizarlo la funciónwithVisible().try(),suppressWarnnings()ysuppressMessages(): Con dichas funciones se puede aplicar el manejo de excepciones.- Véase la documentación sobre las funciones
base::on.exit()ybase::stop().
- Véase la documentación sobre las funciones
base::traceback(): Al momento de tener un error en alguna función, la funcióntracebackpuede ser utilizada para ver los errores en la pila de ejecución.package:lobstr: Este paquete ofrece varias funciones para el manejo de objetos; como la obtención de un identificador de la dirección de memoria que tiene un objeto, su tamaño en bits, obtener en mejor orden el resultado de ciertas funciones; por ejemplo conlobstr::cst()se obtiene el árbol de la pila de llamadas obtenido porbase::traceback(), el tamaño de memoria utilizada en la sesión actual, etc.package:rlang: Las funciones de este paquete están diseñadas para trabajar con ambientes.package:profvisypackage:bench: Cuando se desea optimizar el funcionamiento del código, es común utilizar funciones para evaluar la rápidez de las funciones o rutinas con las que se trabaja; estos paquetes ayudan a realizar un perfilamiento del código y a realizar microbenchmark en funciones pequeñas. Véase el siguiente enlace para más funciones.
varfunction (x, y = NULL, na.rm = FALSE, use)
{
if (missing(use))
use <- if (na.rm)
"na.or.complete"
else "everything"
na.method <- pmatch(use, c("all.obs", "complete.obs", "pairwise.complete.obs",
"everything", "na.or.complete"))
if (is.na(na.method))
stop("invalid 'use' argument")
if (is.data.frame(x))
x <- as.matrix(x)
else stopifnot(is.atomic(x))
if (is.data.frame(y))
y <- as.matrix(y)
else stopifnot(is.atomic(y))
.Call(C_cov, x, y, na.method, FALSE)
}
<bytecode: 0x7fda87e61128>
<environment: namespace:stats>La función anterior calcula la varianza de una variable, en este caso un vector. Véase que al final de la descripción se menciona la palabra bytecode, la cual resulta familiar cuando ya se trabajo en el lenguaje de programación Java. A partir de la versión 2.14 de R, todas las funciones estándar y paquetes fueron pre compilados en bytecode, lo cual otorga rapidez.
- Si el usuario desea compilar alguna función en bytecode lo puede hacer con la función
compiler::cmpfun(). - Si existe la inclusión del compilador bytecode en R ¿Existirá algún método para realizar JIT? Sí.
Ejercicios
Comparar la función del ejercicio 20 con la función
sum()conbench::mark().Obtener la unión, intersección y diferencia del siguiente conjunto de letras.
set.seed(20)
MixLetters <- sample(c(LETTERS, letters),size = 30)Buscar algún data frame del paquete
datasets. Guardar en una variable los primeros 10 registros y en otra los últimos 10.Verifica el siguiente resultado usando la función
integrate().
\[ \int_0^1sin(\pi x)dx = \frac{2}{\pi} \]
- Verifica con la función
integrate()que la famosa función de densidad de una normal integra \(1\) en todo su rango. A continuación se deja dicha función y si se desea basta con usar los parámetros de una normal estándar (\(\sigma = 1\) y \(\mu = 0\)).
\[ f(x) = \frac{1}{\sqrt{2\pi\sigma^2}}e^{-\frac{(x-\mu)^2}{2\sigma^2}} \]
Utilizar la función
bench::mark()para comparar la rapidez entre las funcionesrowSums()yapply(sum)en una matriz numérica.Obtener la suma y producto acumulado de los primeros 100 números naturales.
Dentro del repositorio de este proyecto se encuentra el archivo Data_fake.csv. Este debe cargarse en la sesión actual de R y guardarse como un data frame con el nombre de
Data_fake.Del data frame
Data_fakeobtener por columna el mínimo, máximo y mediana con alguna función de la familia apply en aquellas columnas que sea posible.Usar alguna de las funciones resumen en
Data_fake.Crear una función que acepte un data frame como input y devuelva una lista con dos elementos; el primero será un data frame con solo aquellas variables que sean de tipo categóricas sobre el input y la segunda entrada de la lista de retorno un data frame con las variables numéricas.
Utilizando la función anterior sobre
Data_fake, obtener el data frame con solo variables numéricas y aplicar la función del inciso 45.Análogo a lo anterior, obtener el data frame con solo variables numéricas de
Data_fakey dividir cada variable en 5 intervalos.Utilizar alguna función de la familia apply para aplicar la función
table()sobre cada una de las variables del data frameData_fake.Con los resultados anteriores, obtener aquellos elementos que son más recurrentes.